错误涉及我几天前所发文章“新冠君,你额头的Lab金印怎么洗”的第7部分。
该部分“7.SARS-CoV-2带有实验室载体pShuttle的核苷酸序列INS1378”说:
“SARS-CoV-2的基因序列中,有一个长度为1378的核苷酸序列INS1378,它来自实验室多功能载体pShuttle SN vector。。。”
这一说法参考自美国匹兹堡大学高级研究员詹姆斯·莱昂斯-魏勒 (James Lyons-Weiler)的有关文章。
2020年1月30日,James Lyons-Weiler 在其个人网站发表了一篇题为“On the Origins of the 2019-nCoV Virus, Wuhan, China”的文章,
https://jameslyonsweiler.com/2020/01/30/on-the-origins-of-the-2019-ncov-virus-wuhan-china/
这篇文章还有一篇关联文章:Moderately Strong Confirmation of a Laboratory Origin of 2019-nCoV
https://jameslyonsweiler.com/2020/02/02/moderately-strong-confirmation-of-a-laboratory-origin-of-2019-ncov/
Lyons-Weiler的主要观点包括:
1)最相似的蝙蝠冠状病毒(CoVZC45)与2019-nCoV之间缺乏基因序列同源性;
2)新冠基因组中的核苷酸子序列INS1378与SARS刺突蛋白的序列高度相似;
3)INS1378与pShuttle-SN载体质粒也具有显著的序列相似性,该载体可用于生产SARS疫苗;
4)2019-nCoV非常可能是实验室用来制备SARS疫苗的重组病毒。
在审视、完善“新冠君,你额头的Lab金印怎么洗”一文过程中,我细读了Lyons-Weiler的原文,注意到有网友指出了Lyons-Weiler分析中的错误。在多方查阅相关文章,反复分析、研究正反两方面的看法后,我得出如下综合结论:
1)由Lyons-Weiler文章中的依据,并不能得出INS1378来自实验室载体pShuttle-SN的结论;
2)INS1378是SARS-CoV-2的S1蛋白的一部分,它包含S1蛋白RBD(Receptor Binding Domain,受体结合域)的大部分。
3)SARS-CoV-2的S1蛋白的RBD,克隆自SARS-CoV的S1蛋白的RBD(实际克隆的是一个比RBD要宽的区域,不妨称之为扩展RBD),克隆后的区域还存在进一步的若干基因编辑。SARS-CoV-2的(扩展)RBD,与SARS-CoV的(扩展)RBD,有很高的同源性。Lyons-Weiler观察到的INS1378与SARS-CoV的显著相似性,即来自这种同源性。
4)在将SARS-CoV的(扩展)RBD,克隆到SARS-CoV-2中时,使用了载有SARS-CoV的S1蛋白的实验室载体pShuttle-SN,这就是Lyons-Weiler观察到的,INS1378与pShuttle-SN存在显著相似性的原因。
先谈谈结论1)的理由。
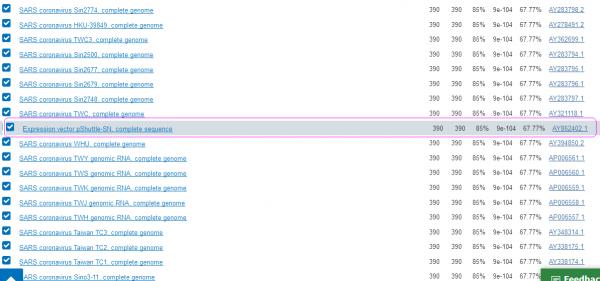
a)INS1378与SARV-CoV刺突蛋白(S蛋白)的序列一致性(可匹配度)并不高,仅为67.77%;它与pShuttle-SN的序列一致性也并不显著,同样仅为67.77%。INS1378与pShuttle-SN,与所有SARV-CoV病毒株基因序列的一致性都是67.77%,对INS1378来说,pShuttle-SN与SARS-CoV并无不同。如果可以说,INS1378来自pShuttle-SN,那么,也可以说INS1378来自SARS-CoV。
下图是用NCBI(National Center for Biotechnology Information)工具Blast得到的INS1378在基因数据库中的搜索匹配结果列表(部分),中间一行是INS1378与“Expression vector pShuttle-SN”的基因序列匹配情况,二者的序列一致性,或者说可匹配度为67.77%。

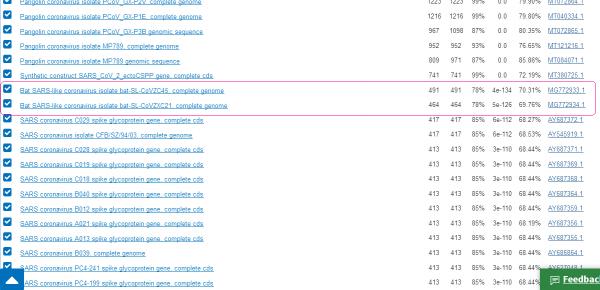
b)INS1378与两种舟山病毒CoVZC45,CoVZXC21的一致性分别为70.31%,69.76%,皆高于其与pShuttle-SN或SARS-CoV的一致性(67.77%),与其说INS1378来自pShuttle-SN,那么,还不如说INS1378来自CoVZC45或CoVZXC21。参见下图。

c)Expression vector pShuttle-SN质粒基因序列共有5607个碱基对,它自编号945开始的一段长为1525的碱基对序列,与SARS-CoV(以病毒株SARS coronavirus BJ162代表)自编号21447(位于SARS-CoV的S蛋白的起始位置21493之前,相距46个碱基对)开始的一段长为1526的碱基对序列高度匹配,在1526个对应位置中,匹配的碱基对有1520个,一致度(匹配度)高达1520/1526=99.61%。下图为比照结果的截图(匹配段序列较长,截图只显示了前面一小部分匹配段)。
Blast Job Title AY862402:Expression vector pShuttle-SN, complete...
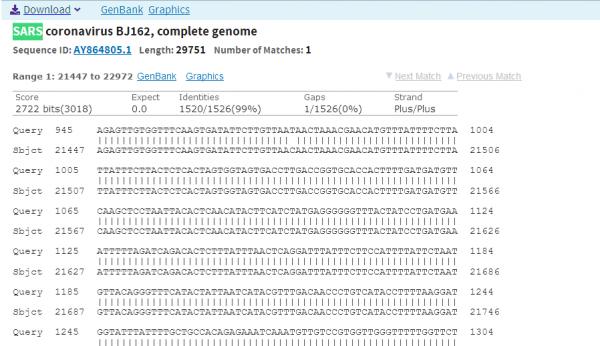
Expression vector pShuttle-SN与SARS-CoV(SARS coronavirus BJ162)基因序列匹配情况
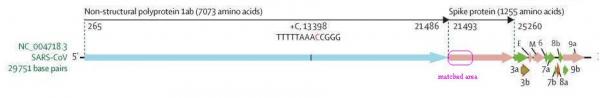
SARS-CoV中,与pShuttle-SN高度匹配区域的位置。
d)Expression vector pShuttle-SN与SARS-CoV二者长为1526的高度匹配区域,包含它们各自与INS1378的最佳匹配区域,因而,INS1378与二者的最佳匹配区域,几乎没有什么不同,这就是INS1378与二者的一致性相同,皆为67.77%的原因。
e)Expression vector pShuttle-SN与SARS-CoV高度匹配的原因是,Expression vector pShuttle-SN中长为1525的序列,本来就是从SARS-CoV的S蛋白中克隆来的。在实验室用SARS-CoV的S蛋白与(缺陷型)腺病毒合成SARS疫苗时,要将SARS-CoV的S1蛋白的一部分(e.g.,from −45 to 1468, SN),克隆到pShuttle质粒中,这一操作得到的一份含有SARS-CoV S1蛋白片断的pShuttle质粒重组克隆体实例,就是Expression vector pShuttle-SN,pShuttle中的S1蛋白片断,就是它与SARS-CoV的高度匹配部分。2004年12月21日,中山大学肿瘤防治中心将Expression vector pShuttle-SN的基因序列,提交至国际基因库GenBank。参见下图。
关于腺病毒SARS疫苗的专利情况及疫苗制备方法,可参见下面两个图。

--- 有一个事实不知大家是否注意到了,早在2003-2004年间,中山大学就已经能够用SARS-CoV的S蛋白与腺病毒骨架来合成人工病毒(疫苗)了。 ---
以上5点说明,就Lyons-Weiler文章中的依据而言,并不能得出“INS1378来自实验室载体pShuttle-SN”这一结论。
那么,SARS-CoV-2基因序列的子序列INS1378与实验室载体pShuttle-SN是不是毫无关系呢?通过对本文后三个结论的讨论可以解答这一问题。
核苷酸(碱基对)序列INS1378在SARS-CoV-2基因序列中的起始序号是21643,终了序号是23020,它正好落在SARS-CoV-2的S1蛋白对应的基因序列内。INS1378在S1蛋白基因序列中的起始位置是95(大致对应第95/3=32个氨基酸),终了位置是1472(大致对应第491个氨基酸),涵盖了大部分的RBD域。参见下面两个图。
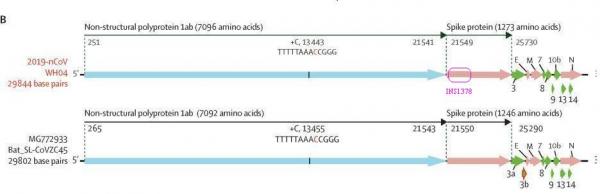
INS1378在SARS-CoV-2的S蛋白中的位置。一个氨基酸(amino acids)对应三个碱基对。
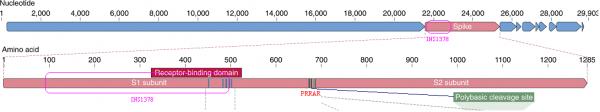
INS1378在SARS-CoV-2的S1蛋白中的位置,及它与RBD的位置关系。
由此,我们可得出本文的结论2)。
2)INS1378是SARS-CoV-2的S1蛋白的一部分,它包含S1蛋白RBD(Receptor Binding Domain,受体结合域)的大部分。
下面看本文结论3)。
3)SARS-CoV-2的S1蛋白的(扩展)RBD,首先克隆自SARS-CoV的S1蛋白的(扩展)RBD,然后在其基础上又作了进一步的基因编辑。也说是说,SARS-CoV-2的(扩展)RBD乃至S1蛋白,与SARS-CoV的(扩展)RBD或S1蛋白,有很高的同源性。Lyons-Weiler观察到的INS1378与SARS-CoV的显著相似性,即来自这种同源性。
这一结论有两个依据。
a)2020年1月21日,来自中国科学院上海巴斯德研究所等单位的徐心恬、陈萍、王靖方等学者在SCIENCE CHINA Life Sciences(《中国科学:生命科学》)上发表了一篇论文,题为“源于武汉爆发的新型冠状病毒的进化及其棘突蛋白对人类传播风险的建模”(Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission)。论文的结论之一是:
尽管新冠与SARS-CoV二者S蛋白的总体同源性较低,但二者的RBD区域却有较高的同源性。
b)2020年1月30日,中国疾控中心、山东第一医科大学的陆柔剑等研究人员在柳叶刀(The Lancet)上联合发表了一篇题为“Genomic characterization and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding”(2019年新型冠状病毒的基因组表征和流行病学:对病毒起源和受体结合的启示)的论文,论文的结论之一是:
虽然2019-nCoV(即SARS-CoV-2)与SARS-CoV的亲缘关系较远,但同源性建模显示,2019-nCoV具有与SARS-CoV相似的受体结合域结构,尽管某些关键残基处存在氨基酸差异。
讨论结论4)。
Lyons-Weiler不仅观察到INS1378与SARS-CoV的显著相似性,同时还观察到INS1378与pShuttle-SN的显著相似性,这又是为什么呢?因为在将SARS-CoV的(扩展)RBD,克隆到SARS-CoV-2中时,使用了载有SARS-CoV S1蛋白的实验室载体pShuttle-SN,pShuttle-SN中的S1蛋白,就是SARS-CoV的S1蛋白的克隆体,所以INS1378与SARS-CoV的相似性,等同于INS1378与pShuttle-SN的相似性。这就是本文的结论4)。
综上所述,Lyons-Weiler所说,SARS-CoV-2基因序列的子序列INS1378来自pShuttle-SN并不准确。实际上,INS1378一部分来自CoVZC45的S1蛋白,一部分来自SARS-CoV的S1蛋白(的扩展RBD),INS1378中还有一些后期的其它基因编辑。也就是说,INS1378只是部分来自SARS-CoV或pShuttle-SN。
虽然Lyons-Weiler对INS1378与pShuttle-SN关系的判断并不准确,但这一判断仍折射出SARS-CoV-2的起源真相。Lyons-Weiler文章中有重要借鉴价值的内容还包括:
a)2019-nCoV进化树分支的bootstrap值(自展值)较低,仅为76,是进化树各分支中最低的。这说明该分支(源于自然进化)的可信度较低。
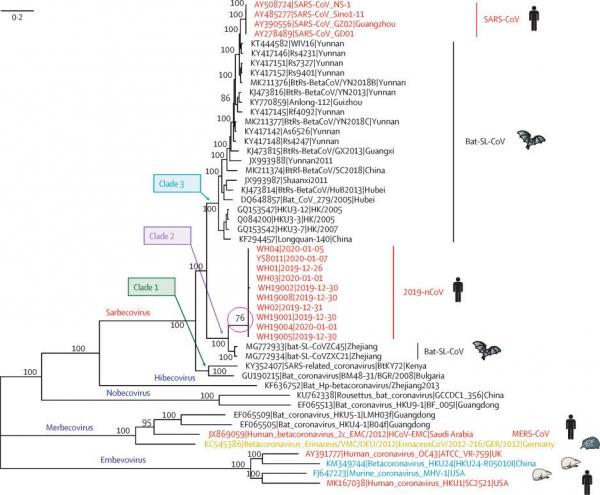
b)2019-nCoV与蝙蝠冠状病毒的基因序列同源性较差,且序列一致性关系不连贯,存在明显的间断gap,这也说明2019-nCoV不太可能由蝙蝠冠状病毒自然进化产生。
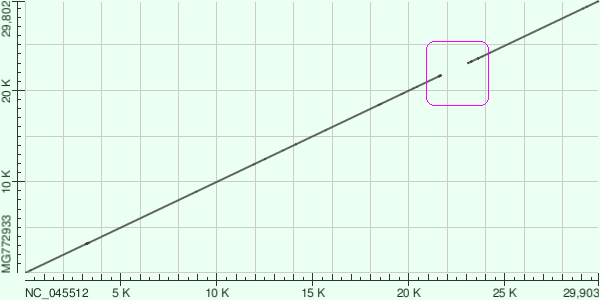
NCBI Blast工具生成的SARS-CoV-2与舟山病毒CoVZC45序列一致性dot plot。
图中有非常明显的间断gap,这一间断gap正好对应着来自SARS-CoV的扩展RBD。
注:NC_045512是SARS-CoV-2的NCBI refseq(登录号),MG772933是CoVZC45的Accession number。
总结一下。“新冠君,你额头的Lab金印怎么洗”一文中的第七道Lab“金印”,其实是第一道Lab“金印”(SARS-CoV-2的RBD克隆自SARS-CoV)的一种欠准确描述。“新冠君,你额头的Lab金印怎么洗”指出的六道实验室“金烙印”,是SARS-CoV-2自然产生论者无法解释,无法面对的,它们揭示了SARS-CoV-2的真正来源。
|
